Mol Cell | 功能相关的基因通过染色质环实现共调控
参与同样生物学过程或组成同一复合物亚单元的基因存在共调控,这一现象在多个物种中保守[1,2],其背后的机制包含基因沿线性基因组的局部聚类等[3,4]。
近日,欧洲分子生物学实验室的Eileen E.M. Furlon团队在Molecular Cell上发表了题为Chromatin gene-gene loops support the cross-regulation of genes with related function的文章,利用Capture-C、DNA原位荧光杂交(FISH)、遗传干扰等技术,他们发现许多同源基因会形成一个染色质环(loop)来维持其共表达,这一染色质环的破坏会使其基因表达改变,并且进一步干扰其共调控的能力。

作者首先对果蝇胚胎发生三个不同阶段进行深度的原位HiC测序,分析发现不同胚胎阶段拓扑相关结构域(TADs)的基因组架构是类似的,在TADs内部甚至多个TADs中存在许多非常高频率的loops,作者在三个时间点分别鉴定出了475,633和962个loops,特别地,作者发现这其中包含许多长程(跨越几个Mb)的loops。进一步地,他们探究了形成loop的相关基因,发现有45%的loops在同源基因中形成,包括许多发育基因、编码免疫球蛋白跨膜蛋白的基因等,非同源基因则涉及同一生物学途径。接下来,作者挑选了DNA原位荧光杂交中的三个区域进行单细胞水平的HiC比较,同样发现了高频的基因-基因loop。
为评估loop互作对于基因表达调控的影响,作者对scyl-chrb进行遗传干扰,分别破坏scyl、loop anchor(LA)、scylLA和整个chrb基因,Capture-C和FISH检测结果显示,删除scyl不影响成环,删除LA不影响外部loop,但使LA、scyl及chrb的互作丧失。进一步的分析显示,当loop完整时,scyl和chrb对彼此的表达是负调控关系,而当loop被破坏,即使两基因保持完整,二者也不再具备相互调控的能力,这提示基因-基因间环经常直接或间接参与交叉调控彼此的共表达基因。
tsh-tioloop的遗传干扰试验进一步证实了这一点,作者使用不同策略删除tsh-tio的不同元件,发现删除外部LA会极大减少tsh-tioloop的数量,但并不导致胚胎前端两基因的异常表达,说明这一loop对于基因调控不是必需的。而在胚胎后端,在两基因都活跃的情况下破坏tsh-tioloop会导致二者的表达下调,当LA和tsh基因被移除时,tio表达会显著上调,这说明顺式元件cis中loop的破坏会干扰其中一个基因交叉调控另一基因的能力。长程loops中,GluRIA-GluRIB的存在被证实,同样通过遗传敲除,作者证实了两基因对彼此表达的调控,特别地,他们发现这一调控在loop存在时进一步增强,破坏环能够影响基因表达,干扰其共调控。
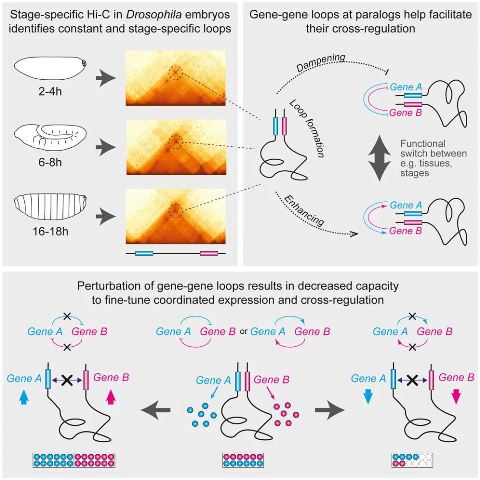
综上,文章发现染色质基因-基因环能够作为结构骨架,在不同情境下以不同方式实现并维持对功能相关基因共表达的精细调控。
(来源:BioArt)
参考文献:
1. Williams, E.J.B., and Bowles, D.J. (2004). Coexpression of neighboring genes in the genome of Arabidopsis thaliana.Genome Res.14, 1060–1067. https://doi.org/10.1101/gr.2131104.
2. Ribeiro, D.M., Rubinacci, S., Ramisch, A., Hofmeister, R.J., Dermitzakis, E.T., and Delaneau, O. (2021). The molecular basis, genetic control and pleiotropic effects of local gene co-expression.Nat. Commun.12, 4842. https://doi.org/10.1038/s41467-021-25129-x
3. Hurst, L.D., Pa´l, C., and Lercher, M.J. (2004). The evolutionary dynamics of eukaryotic gene order.Nat. Rev. Genet. 5, 299–310. https://doi.org/10. 1038/nrg1319.
4. Soler-Oliva, M.E., Guerrero-Martı´nez, J.A., Bachetti, V., and Reyes, J.C. (2017). Analysis of the relationship between coexpression domains and chromatin 3D organization.PLoS Comput. Biol.13, e1005708. https://doi.org/10.1371/journal.pcbi.1005708.